京都大:iPSでアルツハイマー病関連遺伝子を特定(動画):
Kyoto Univ: Identifies genes related to Alzheimer’s disease with iPS:
京都大学:用 iPS 识别与阿尔茨海默病相关的基因:
京都大
井上治久教授(神経内科学)「アルツハイマー病患者から作ったiPS細胞」を使い、
「アルツハイマー病に関わる複数の遺伝子」を特定した。
研究チームが21日までに発表した。
これらの遺伝子を標的とし、早期診断や治療薬の開発につなげる。
アルツハイマー病:
アルツハイマー病は、
「脳内に特定のタンパク質が過剰に蓄積されること」が原因とされる。
今回、病気のうち9割以上を占め、家族に病歴がない「孤発性」について調べた。
血液細胞の遺伝子分析:
研究チームは、
患者102人の血液細胞から作ったiPS細胞を、脳の神経細胞に成長させ、病気を細胞レベルで再現した。
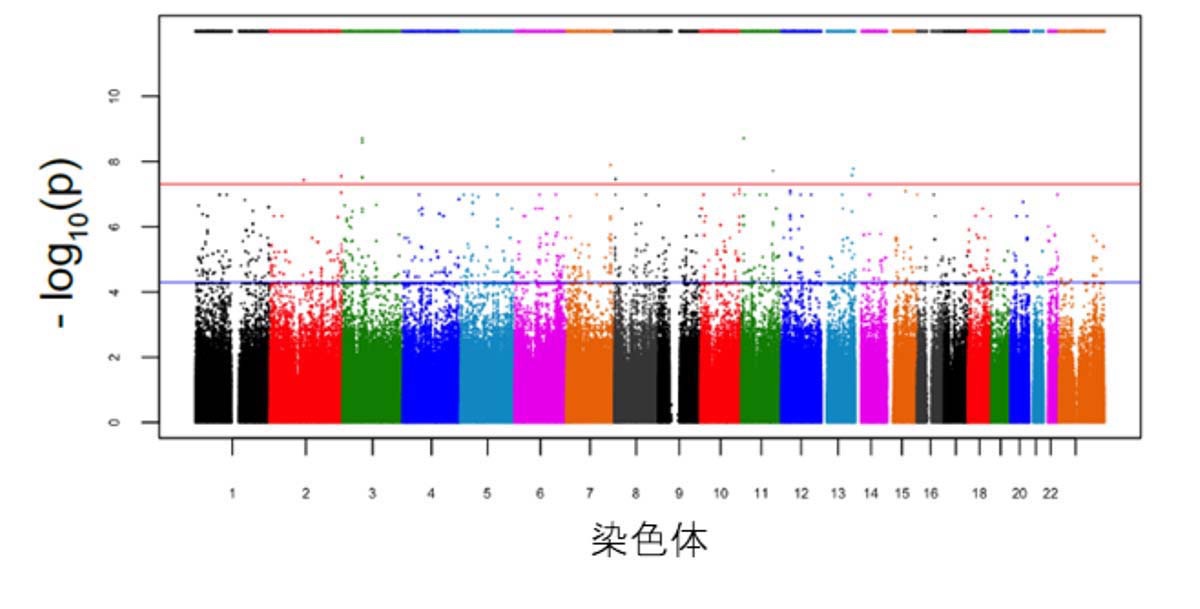
24の遺伝子を特定:
細胞の遺伝子の働きなどを分析。
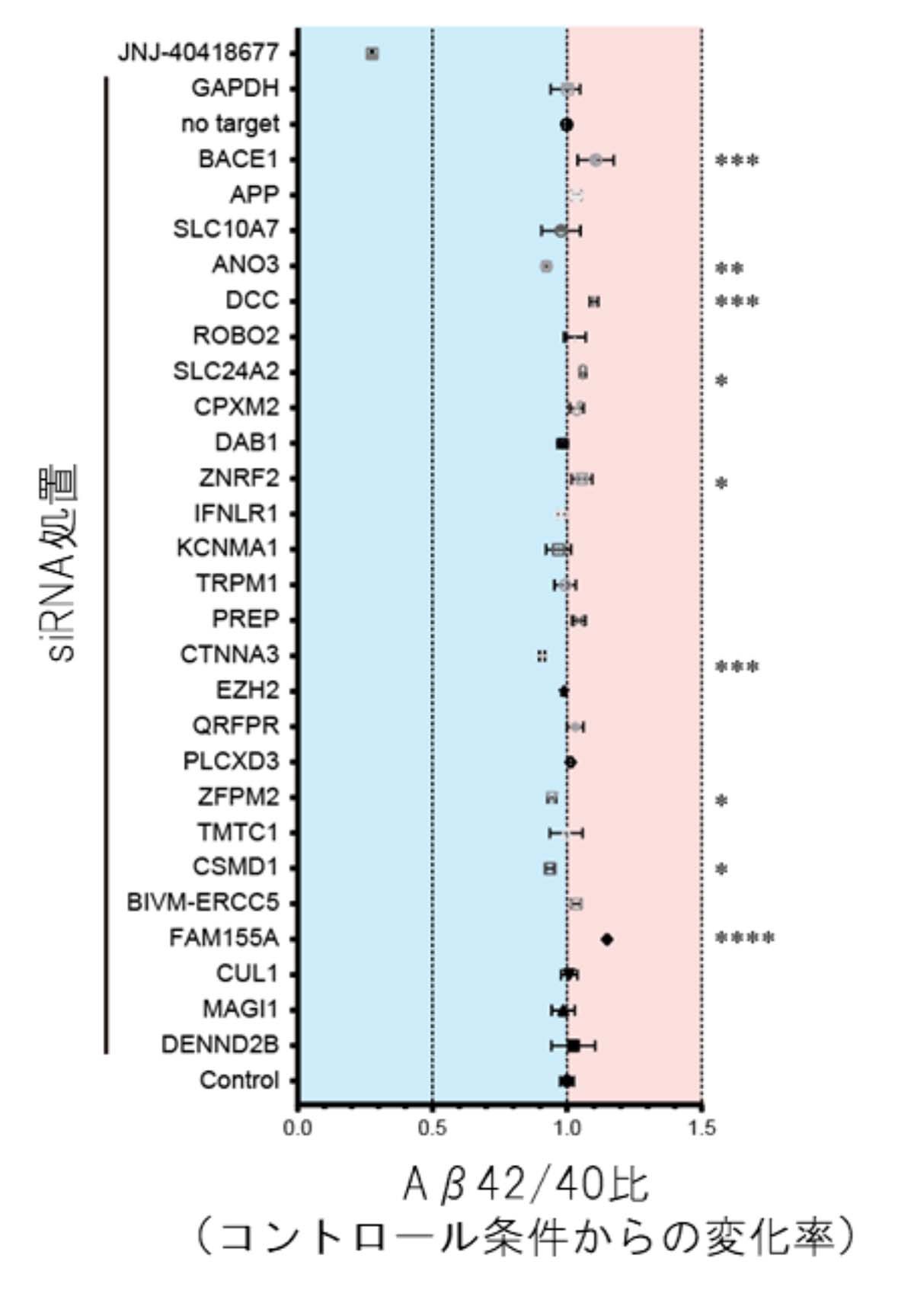
「特定タンパク質に関係するとみられる24の遺伝子」を特定した。
さらに、
「このうち8の遺伝子は特定タンパク質の量の調節に関わること」も分かった。
高精度予測モデル:
特定した遺伝子の情報や臨床データなどを活用する。
個人の遺伝情報から病気の発症を、高精度で予測するモデルを構築する。
日本経済新聞
https://www.nikkei.com/article/DGXZQOUE219O80R20C22A2000000/
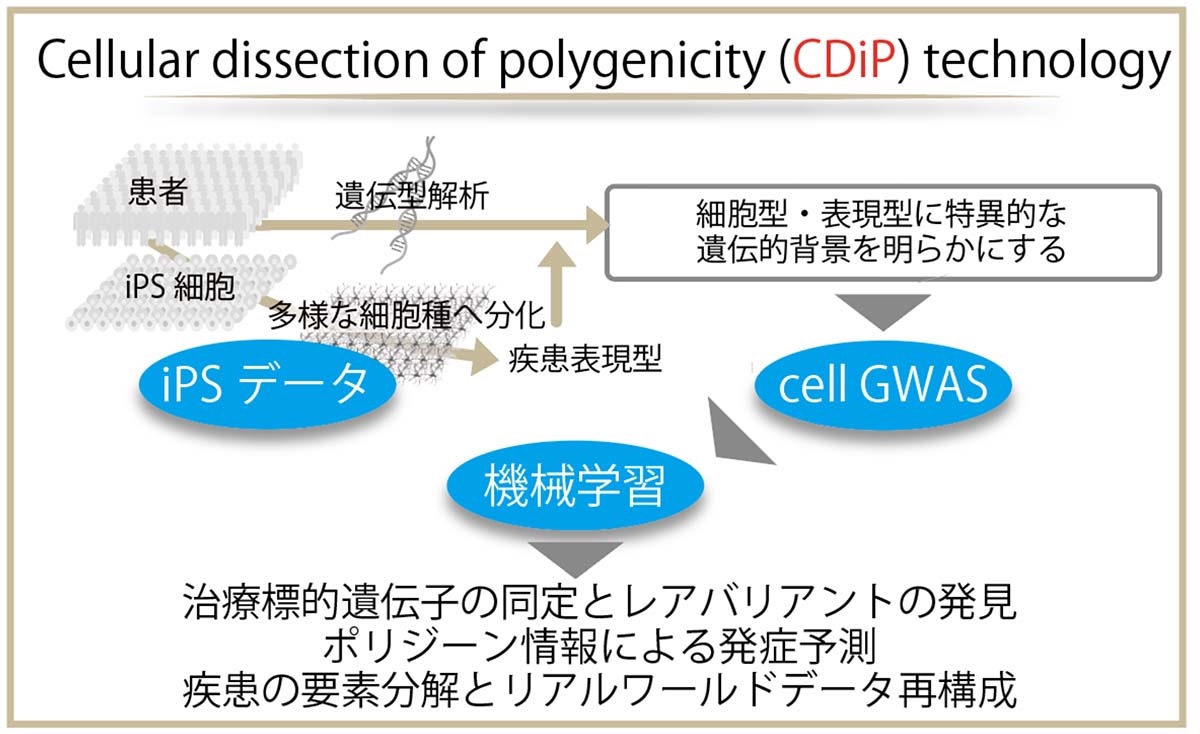
iPSコホートと機械学習を用いたアルツハイマー病再構成
-CDiPテクノロジーによる無病社会に向けた孤発性高齢疾患の解読-
CiRA | 京都大学 iPS細胞研究所
https://www.cira.kyoto-u.ac.jp/j/pressrelease/news/220218-010000.html
Dissection of the polygenic architecture of neuronal Aβ production using a large sample of individual iPSC lines derived from Alzheimer’s disease patients
Nature Aging
Abstract
Genome-wide association studies
have demonstrated that polygenic risks shape Alzheimer’s disease (AD).To elucidate the polygenic architecture of AD phenotypes at a cellular level,
we established induced pluripotent stem cells from 102 patients with AD, differentiated them into cortical neurons and conducted a genome-wide analysis of the neuronal production of amyloid β (Aβ).
Using such a cellular dissection of polygenicity (CDiP) approach,
we identified 24 significant genome-wide loci associated with alterations in Aβ production,
including some loci not previously associated with AD, and confirmed the influence of some of the corresponding genes on Aβ levels by the use of small interfering RNA.
CDiP genotype
sets improved the predictions of amyloid positivity in the brains and cerebrospinal fluid of patients in the Alzheimer’s Disease Neuroimaging Initiative (ADNI) cohort.Secondary analyses of exome sequencing data from the Japanese ADNI and the ADNI cohorts
focused on the 24 CDiP-derived loci associated with alterations in Aβ led to the identification of rare AD variants in KCNMA1.